 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2011 年, 第 9 卷, 第 92 篇 doi: 10.5376/mpb.cn.2011.09.0092
收稿日期: 2011年06月17日 接受日期: 2011年07月05日 发表日期: 2011年07月12日
引用格式(中文):
刘娜等, 2011, 苹果茎痘病毒梨分离物外壳蛋白基因的克隆与分子变异分析, 分子植物育种(online) Vol.9 No.92 pp.1653-1661 (doi: 10.5376/mpb. cn.2011.09.0092)
引用格式(英文):
Liu et al., 2011, Cloning and Sequencing and Molecular Variability of Apple stem pitting virus CP Gene from Pear, Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding) Vol.9 No.92 pp. 1653-1661 (doi: 10.5376/mpb.cn.2011.09.0092)
本研究利用RT-PCR技术扩增了来自新疆地区的库尔勒香梨、鸭梨的苹果茎痘病毒外壳蛋白基因,克隆测序结果表明,ASVP CP基因全长1 194 bp,编码397个氨基酸,分子量约42 KDa。与GenBank上公布的25个ASVP CP基因编码的氨基酸序列的同源率为70%左右。多重氨基酸序列比对结果显示,aa1-177之间变异较大,为高变区,而后130个氨基酸高度保守。对CP基因位点扫描发现,CP基因的活性和识别位点主要分布在高度保守区域,位点包括:Cys位于aa292,ProProXXTrp位于aa309-313,AryXPheAspPhe位于aa325-329及Pro位于aa349。系统进化分析结果表明,来自世界各地的ASPV分离物可分为3大类群,分别为:第一类群是以苹果为寄主的类群,第二类群是以梨为寄主的类群,第三类群是以新疆栽培的梨品种为寄主的类群。呈现一定的寄主相关性。
苹果茎痘病毒(Apple stem pitting virus, ASPV)是凹陷病毒属(Foveavirus) 的典型代表,为线状弯曲病毒(Martelli and Jelkmann, 1998)。ASPV是一种严重危害果树生产的潜隐性病毒,主要侵染苹果和梨,广泛分布于世界各地的苹果和梨种植区。由ASPV引起的梨树病害有梨石痘病(Pear stony pit)、梨茎痘病(Pear stem pitting)、梨脉黄病(Pear vein yellow)等多种病害,并常伴随苹果褪绿叶斑病毒(Apple chlorotic leaf spot virus, ACLSV)和苹果茎沟病毒(Apple stem groove virus, ASGV)进行复合式侵染,引起果实品质变劣,产量降低,造成严重的经济损失。
目前在GenBank上登录ASPV分离物全基因组序列仅有4条,分别为德国分离物PA66,登录号为NC_003462 (Jelkmann, 1994);日本分离物IF38,登录号为AB045371 (Yoshikawa et al., 2001);中国分离物PR1,登录号为EU095327和印度分离物Palampur,登录号为FR694186。而有关ASPV CP基因的完整编码序列或部分序列已有来自不同国家和地区的很多报道。现有的基因组序列和部分序列已表明ASPV不同分离物存在着复杂的分子变异。因此,非常有必要系统开展ASPV的分子变异、遗传进化和株系分化研究。但目前国内外报道的ASPV全长基因组序列仅有4条,利用这4条序列开展ASPV的遗传进化及分子变异研究尚存在一定局限性。Nemchinov等(1998)采用PCR技术检测了不同寄主的ASPV,同时对不同分离物进行了序列分析; Schwarz和Jelkmann (1998)检测了欧洲梨和苹果分离物的ASPV,进行了外壳蛋白基因的序列分析。分析结果均表明ASPV不同分离物间存在变异。然而国内外关于ASPV CP基因分子变异及遗传进化研究方面系统报道较少,因此,利用外壳蛋白基因序列开展分子变异研究十分必要。通过了解病毒变异规律及株系分类情况,明确病毒变异特点对更好的认识病毒和控制病毒具有重要意义。本研究分别从新疆库尔勒地区香梨园中采集库尔勒香梨、鸭梨、砀山梨等主要栽培品种,通过对ASPV CP基因不同分离物的克隆及详细的序列分析,以明确其分子变异规律和分类特点。
1结果与分析
1.1ASPV CP基因的RT-PCR扩增
以感染ASPV的库尔勒香梨KL1、KL9、鸭梨Y3及砀山梨的枝条韧皮部为试材,将提取的总RNA反转录合成cDNA第一链后,利用设计的引物PS/PA 进行RT-PCR扩增,KL1、L9、鸭梨Y3分别扩增出了预期片段,大小为1 392 bp,包含了完整的CP基因。感染ASPV的砀山梨没有扩增出目的片段(图1)。
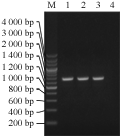 图1 ASPV CP基因的RT-PCR检测 Figure 1 Detection of ASPV CP gene by RT-PCR |
1.2 ASPV外壳蛋白基因的克隆与鉴定
RT -PCR产物经回收纯化后连接到PMD19-T带有抗氨苄青霉素(Ampr)抗性的载体上,热击转化大肠杆菌DH5α。通过含有Amp、X-gal和 IPTG的LB培养基37℃培养12 h~16 h,进行蓝白斑筛选。挑取白色菌落,振荡培养后,用裂解法小量提取重组质粒DNA,做重组质粒的PCR及酶切鉴定(图2)。将鉴定正确的质粒菌液送往上海生工生物技术有限公司测序。
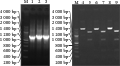 图2 重组质粒的PCR, 酶切鉴定 Figure 2 Identification of recombined plasmid by PCR and Restriction enzyme |
1.3 ASPV外壳蛋白基因的序列测定
测序分析结果表明,3个分离物(KL1、KL9、Y3)的ASPV外壳蛋白基因片段大小均为1 194 bp,编码397个氨基酸,分子量约为42 kDa。将序列提交至GenBank,登录号分别为JF946775、JF946773、JF946774。ASPV CP-KL1中含有316个A,292个T,293个G和293个C,各碱基在CP基因中的百分含量分别为26.3%、24.5%、24.5%和 24.5%;ASPV CP-KL9中含有314个A,291个T,296个G和293个C,各碱基在CP基因中的百分含量分别为26.5%、24.4%、24.6%和 24.5%;ASPV CP-Y3中含有320个A,291个T,288个G和295个C,各碱基在CP基因中的百分含量分别为26.8%、24.4%、24.1%和 24.7%;三条序列的碱基变化显示,A和G的变异较大,从而引起编码的氨基酸序列发生变化。
1.4 ASPV外壳蛋白基因的序列分析
到目前为止,已经报道的ASPVCP基因完整编码序列25条(表1)。通过DNAMAN、Clustalx及MEGAR 4.0等软件分析,对本研究得到的3个ASPV CP基因的分离物(KL1, KL9, Y3)同已报道的ASPV CP基因分离物的全序列进行同源性、系统进化及分子变异分析。
 表1 ASPV CP基因分离物登录号及地理来源 Table 1 Accession number and geographical origin of the isolate of ASPV CP gene |
1.4.1同源性分析
KL1 与KL9、Y3,KL9与Y3的核苷酸序列同源率分别为90.5%、90.2%和96.7%;氨基酸序列的同源率分别为93.0%、92.7%和 97.3%;与GenBank上公布的20多个ASPV分离物的CP基因氨基酸序列进行比对分析,不论按其地理分布还是根据寄主范围,三个分离物与其比对的同源率均为68.2%~74.4% (表2)。
 表2 KL1, KL9, Y3 CP基因与ASPV其它分离物氨基酸同源率比对 Table 2 Identities of CP amino acid sequences KL1, KL9 and Y3 with other isolates |
氨基酸序列多重比对结果表明,aa1-177出现较大变异,为高变区,其中在aa28-46,NC_003462多了18个氨基酸,HM352767在 aa37-50多了14个氨基酸,KL1、KL9及Y3在aa129-130处发生缺失(图3)。而后130个氨基酸则为高度保守区域。研究结果表明, ASPV CP基因的变异在每个分离物中普遍发生,没有呈现一定的规律性。对CP基因的氨基酸序列位点扫描分析发现,CP基因识别位点主要分布在高度保守区,位点分别为:Cys位于aa292,ProProXXTrp位于aa309-313,AryXPheAspPhe位于aa325-329和Pro位于 aa349,这些位点是外壳蛋白基因的活性和识别位点。
.png) 图3 ASPV CP基因氨基酸序列多变区多重比对 Figure 3 Alignment of the amino acid sequences of high high variability domain of coat protein ASPV |
1.4.2系统进化分析
我们对3个ASPV新疆分离物CP基因和GenBank上发布的25个ASPV分离物CP基因的氨基酸序列构建了系统进化树(图4),结果显示25个 ASPV分离物分成明显的3个类群,Ⅰ类群是以苹果为寄主,在该类群中有4个进化分支,AB045371 (日本Japan)、HM125157 (中国China)、AF491931 (波兰Poland)、DQ003336 (捷克Czech Republic)及AF491930 (波兰Poland)形成紧密的一簇,构成一个分支;FJ619188 (波兰Poland)与HM125156 (中国China)构成第二个分支;AF438521 (波兰Poland)、FJ619187 (中国China)、HM125158 (中国China)、FN430678 (印度Indian)、FR694922 (印度Indian)形成一簇,构成第三分支,EU314950 (中国China)、AF438522 (波兰Poland)和AF495382 (波兰Poland)构成了第四分支。Ⅱ类群是以梨为寄主,由四个分支构成,第一分支由HM352762 (中国China)和AF345892 (波兰Poland)构成;第二分支由EU708018 (中国China)和AF491929 (波兰Poland)构成;第三分支由AF345893 (波兰Poland)和NC_003462 (德国Germany)构成;AY572458 (巴西Brasil)、AF345894 (波兰Poland)和AF345895 (波兰Poland)形成一簇,构成第四分支。ASPV-KL1、ASPV-KL9及ASPV-Y3构成第Ⅲ类群,寄主为新疆栽培的梨品种。
 图4 基于ASPV CP基因氨基酸序列构建系统树 Figure 4 Phylogenetic tree of ASPV isolates constructed with the complete coat protein-coding aa sequences |
2讨论
本研究扩增的ASPV分离物的CP基因分别来自库尔勒香梨和鸭梨。由于ASPV存在复杂的变异,会对ASPV CP基因的扩增造成一定困难。本实验使用的PS/PA是有效扩增CP基因完整编码区的引物。通过大量样品扩增发现,库尔勒香梨、鸭梨、砀山梨这三个品种的样品中,库尔勒香梨和鸭梨均扩增出了预期目的片段,测序结果证明是ASPV CP基因。本研究扩增出了库尔勒香梨KL1和KL9及鸭梨Y3的3个分离物CP基因,基因片段大小均为1 194 bp,编码397个氨基酸,分子量约为42 kDa。将序列提交至GenBank,登录号分别为JF946775、JF946773、JF946774。而砀山梨的6个样品均没有扩增出目的片段,但用ASPV复制酶保守区引物扩增出了目的片段,并通过目的片段测序证明这些样品均感染了ASPV。因此,应该在不同生态环境和地理区域采集更广泛的寄主研究外壳蛋白基因的分子变异,以便能系统的了解ASPV外壳蛋白的变异特征及株系分化情况。
实验结果显示,我们扩增得到的3个ASPV分离物的CP基因与GenBank上登录的序列的氨基酸的同源率一般均在70%左右。aa1-177氨基酸序列出现较大变异,为高变区,而后130个氨基酸高度保守。外壳蛋白在保护病毒核酸的同时,在病毒侵染过程中还起着识别寄主的作用,而蛋白质功能位点往往是由较短的序列片段组成,此区域序列一般具有较强的保守性(Meng et al., 1998; Hull, 2001)。对其氨基酸序列位点扫描发现,CP基因的识别位点主要分布在高度保守区,分别为:Cys位于aa292,ProProXXTrp位于 aa309-313,AryXPheAspPhe位于aa325-329及Pro位于aa349,这些位点是外壳蛋白基因的活性和识别位点,可能还执行其它重要的生理功能。因此,有关位点及变异区域的研究,对搞清ASPV CP基因的结构功能及其变异特征具有重要的意义。
分子生物学技术在植物病毒分类及病毒株系鉴定中的应用,使得界定植物病毒的地位或株系种类更具准确性和科学性(Li et al., 2006)。随着分子生物学技术的不断进步,利用病毒序列来研究病毒的演化已成为可能,用其构建系统进化树可以准确了解病毒株系的亲缘关系及演化趋势(Chachulska et al., 1997)。对ASPV来说,由于目前报道的全长基因组序列仅有4条,对该病毒的分类及株系分化带来了一定困难,而病毒的CP基因在寄主症状、病毒长距离和细胞间运转、病毒的介体传播等方面起着重要作用,同时,病毒间CP基因的同源性比较分析是病毒株系划的主要依据之一,因此,本实验对3个新疆分离物的 CP基因与GenBank上登录的25个完整的CP基因氨基酸序列进行了系统进化分析,结果显示,这些分离物分类聚集的趋势十分明显,呈现一定的寄主相关性,该结果与李丽丽等(2010)报道的结果相同。本试验研究结果显示,28个分离物共分为3个类群,一是以苹果为寄主的类群,二是以梨为寄主的类群,三是以新疆栽培的梨品种为寄主的类群。图4显示,新疆栽培的梨品种自成一个类群,第一和第二类群中小的进化分支的形成并不具有一定的规律性,这可能是由于生态环境的改变,引起了病毒外壳蛋白基因的变异。从表1可以看出,25个分离物大多为中国和波兰分离物,这给进化关系的分析带来一定局限性。病毒也同其它生物一样,由于自然选择压力的存在,当自然环境改变时,为了适应环境而不断地发生变异,基因组结构和序列能真实反映植物病毒的变异及进化关系(古勤生等, 2008),如能将不同地理来源,不同气候条件的分离物的CP基因与基因组的其它序列信息结合起来分析,则会使ASPV分类更加全面。
3材料与方法
3.1材料
3.1.1植物材料
本实验所用植物材料均采自新疆库尔勒沙依东园艺场香梨园,采集库尔勒香梨、鸭梨和砀山梨的一、二年生枝条,并放入4℃保存备用。
3.1.2主要试剂和菌株
Taq DNA聚合酶购自广东东盛生物科技有限公司;Ribonuclease Inhibior、dNTPs、IPTG、X-Gal购自上海生工生物技术有限公司;DNA Marker D514A、PMD19-T vector均购自大连宝生物有限公司;反转录酶M-MLV、限制性内切酶购自Fermentas;其它常规试剂均为国产分析纯。菌株E. Coli DH5α由石河子大学农学院果树生物技术实验室保存。
3.1.3引物设计
根据GenBank上已提交的ASPV全长基因组序列(登录号: NC_003462),通过Primer 5.0软件设计扩增CP基因的全序列引物。引物序列为:上游引物(PS) 5'-CCCATTAGGTTAGGGTGTAGTTGCT-3' (位于nt7829-7853);下游引物(PA) 5'-ATGAAAGAAACACACACATAGCCGC-3' (位于nt9249-9273)。引物由上海生工生物技术有限公司合成。
3.2方法
3.2.1总RNA的提取
利用皮层为材料提取总RNA,具体参考何梅等(2004)库尔勒香梨总RNA快速提取方法。
3.2.2苹果茎痘病毒的RT-PCR扩增
cDNA第一链的合成:反转录体系为25 μL,含总RNA 5 μL、DEPC水 9.5 μL、反向引物 1 μL;70℃水浴5 min,冰浴5 min;再依次加入M-MuLV 5× buffer (Mg2+ 40 mmol/L) 5 μL、dNTPs (10 mmol/L) 2.5 μL、RNasin (40 U/μL) 1 μL;42℃水浴5 min,最后加入M-MuLV 1 μL。反应条件为42℃水浴 1 h,92℃灭活4 min,然后置于冰上进行PCR反应或-20℃保存待用。
PCR扩增PCR反应体系:10× Buffer 2.0 μL (含Mg2+)、dNTP 0.4 μL、引物PS/PA (20 μmol/L)各1 μL、cDNA 2.0 μL、Taq聚合酶0.2 μL,总体积20 μL。PCR扩增程序为:94℃预变性4 min,32次循环:94℃ 45 s,57℃ 45 s,72℃ 1 min,最后72℃ 延伸7 min。
3.2.3 PCR产物电泳检测
取上述PCR产物5 μL,用1.2%的琼脂糖凝胶,溴化乙锭(EB)染色,进行电泳鉴定,电泳缓冲液为1× TAE,电压150 V,紫外灯下观察实验结果并照相记录。
3.2.4目的片段克隆、测序和序列分析
PCR产物经切胶回收纯化后与PMD19-T载体连接,转化大肠杆菌DH5α,通过抗性筛选,将质粒PCR、酶切鉴定都正确的插入片段的菌液寄往上海生工生物技术有限公司进行测序。测序结果同源性分析采用DNAMAN软件,氨基酸的多重比对使用CLUSTAL-X (version 1.81)分析软件,系统进化树采用CLUSTAL-X (version 1.81)和MEGA (version 4.1)分析软件。
作者贡献
刘娜是本研究的实验设计和实验研究的执行人,同时完成数据和试验结果分析,论文初稿的写作;牛建新是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究由国家自然科学基金资助项目(30360066);国家科技攻关计划引导项目(2003BA546C);石河子大学自然科学与技术创新项目 (ZRKX200707)的资助。作者感谢两位匿名的同行评审人的评审建议和修改建议。本文中提到了我们实验中涉及的有关试剂供应商和测序服务商,这并非我们为这些试剂供应商和测序服务商的产品和服务提供推荐或背书。
参考文献
Chachulska A.M., Chrzanowska M., Robaglia C., and Zagorski W., 1997, Tobacco veinal necrosis determinants are unlikely to be located within the 5' and 3'-terminal sequences of the potato virus Y genome, Arch. Virol., 142(4): 765-779 http://dx.doi.org/10.1007/s007050050117
Gu Q.S., Tian Y.P., Peng B., Liu L.F., Deng C.L., Liang X.M., and Meng J., 2008, Molecular analysis of three cucurbit viruses based on coat protein gene sequences, Zhiwu Bingli Xuebao (Acta Phytophylacica Sinca), 38(4): 357-363 (古勤生, 田延平, 彭斌, 刘丽锋, 邓丛良, 粱新苗, 孟娟, 2008, 基于为壳蛋白基因序列对3种葫芦科作物病毒的分子分析, 植物病理学报, 38(4): 357-363)
He M., Li H.S., Zhao Y., Niu J.X., and Ma B.G., 2004, A method of rapidly extracting total RNA from Korla Aroma pear, Shihezi Daxue Xuebao (Journal of Shihezi University (Natural Science)), 22(6): 474-476 (何梅, 李海生, 赵英, 牛建新, 马兵钢, 2004, 库尔勒香梨总RNA快速提取方法, 石河子大学学报(自然科学版), 22(6): 474-476)
Hull R., ed., 2001, Matthews’ plant virology, Fourth Edition, Academic Press, London and California, pp.758
Jelkmann W., 1994, Nucleotide sequences of apple stem pitting virus and of the coat prote in gene of a similar virus from pear associated with vein yellows disease and their relationship with potex- and carlaviruses, Journal of Virology, 75(7): 1535-1542
Li L.L., Dong Y.F., Zhang Z.P., Zhang Z.H., Fan X.D., and Pei G.Q., 2010, RT-PCR detection and molecular variability of apple stem pitting virus, Yuanyi Xuebao (Acta Horticulturae Sinica), 37(1): 9-16 (李丽丽, 董雅凤, 张尊平, 张志宏, 范旭东, 裴光前, 2010, 苹果茎痘病毒RT-PCR检测及分子变异分析, 园艺学报, 37(1): 9-16)
Li N., Wang X.F., Zhou G.H., and Dong J.G., 2006, Molecular Variability of the Coat protein gene of Potato virus Y from Tobacco in China, Acta Virologica, 50(2): 107-113
Martelli G.P., and Jelkmann W., 1998, Foveavirus, a new plant virus genus, Arch. Virol., 143(6): 1245-1249 http://dx.doi.org/10.1007/s007050050372
Meng B.Z., Pang S.Z., Forsline P.L., McFerson J.R., and Gonsalves D., 1998, Nucleotide sequence and genome structure of grapevine rupestris stem pitting associated virus-1 reveal similarities to Apple stem pitting virus, Journal of General Virol., 79(8): 2059-2069
Nemchinov L., Hadidi A., and Faggioli F., 1998, PCR-detection of Apple stem pitting virus from pome fruit hosts and sequence variability among viral isolates, Acta Hortic., 472: 67-73
Schwarz K., and Jelkmann W., 1998, Detection and characterization of European Apple stem pitting virus isolates of apple and pear by PCR and partial sequence analysis, Acta Hortic., 472: 75-85
Yoshikawa N., Matsuda H., Oda Y., Isogai M., Takahashi T., Ito T., and Yoshida K., 2001, Genome heterogeneity of Apple stem pitting virus in apple trees, Acta Hortic., 550: 285-290